Aktuelle Beiträge rund um die Bioökonomie
-
Projekt ProGrün - 30.12.2023

Tierfutter enthält häufig Protein aus Soja, dessen Import mit großen Umweltschäden einhergeht. Im Projekt ProGrün entwickeln Forschende der Uni Hohenheim, u. a. die AG von Prof. Dr. Andrea Kruse, ein skalierbares technologisches Verfahren, um Proteine aus Grünlandschnitt zu extrahieren und aus diesen nachhaltig erzeugtes Tierfutter herzustellen. Sonstige anfallende Nebenströme werden in der Bioraffinerie zu hochwertigen Endprodukten aufgewertet.
-
Alternative Baumaterialien: Myzelwerkstoffe - 20.12.2023

Für die meisten von uns sind Pilze nur Nahrungsmittel – eventuell auch noch Krankheitserreger. Ein Fehler, denn diese erstaunlichen Lebewesen sind noch zu viel mehr imstande: Sie wachsen auf pflanzlichen Reststoffen aller Art und verbinden diese zu flexiblen oder festen Formstücken, aus denen neue nachhaltige und wirtschaftlich attraktive Produkte wie Leder- und Styroporersatz oder Baustoffe werden können.
-
Fassadenbegrünung mit Niederschlagsretention - 19.12.2023

Aufgrund der engen Bebauung und der damit einhergehenden Bodenversiegelung werden für Stadtbewohner sowohl Hitze als auch Starkregen immer mehr zum Problem. Forschende der Deutschen Institute für Textil- und Faserforschung (DITF) in Denkendorf haben neuartige Living Walls entwickelt, die nicht nur zur Umgebungskühlung beitragen, sondern dank ihrer Wasserretentionseigenschaften auch einen Überschwemmungsschutz bieten.
-
Nachhaltige Klebstoff-Alternative - 18.12.2023
Kunststoff ist überall um uns – leider immer noch. Die Suche nach Nachhaltigerem ist in vollem Gang. Allerdings braucht es bisher nicht oder nur schwer biologisch abbaubaren Klebstoff, um Alternativen aus Holz oder Stroh zu verbinden – keine wirklich umweltfreundliche Lösung. Fraunhofer-Forschende arbeiten nun an einem insekteninspirierten Holzklebstoff, mit dem verklebte Holzprodukte sowohl widerstandsfähig als auch biologisch abbaubar sind.
-
Voll-Enzym-Hydrogele im Einsatz - 13.12.2023
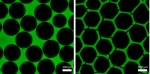
Konventionelle chemische Syntheseprozesse verbrauchen viel Energie und umwelt-schädigende Lösungsmittel. Die Arbeitsgruppe von Prof. Dr. Christof Niemeyer vom Karlsruher Institut für Technologie hat aus vernetzten Enzymen poröse, feste Schäume generiert, die die Produktion hochwertiger Verbindungen unter deutlich umwelt-schonenderen Bedingungen erlauben. Die neuartigen Biokatalysatoren sind zudem extrem widerstandsfähig und lange haltbar.
-
BIOPRO Podcast Folge 8 - 29.11.2023

In dieser Folge beschäftigen wir uns mit den Themen Vertical Farming und Phytopharmaka und erörtern, was das eine mit dem anderen zu tun hat. Wir sprechen mit Dr. Reinhard Bott und seinem Kollegen Daniel Trippe von der Vertical Farm Tech GmbH. Dr. Bott ist auch in der Fachinitiative Phytopharmaka und dem Netzwerk Vertical Farming aktiv, welche von der BIOPRO begleitet werden. Damit ist er der perfekte Kandidat, um unsere Fragen zu beantworten.
-
Neueste Forschung zeigt Wirksamkeit von Biotensiden - 28.11.2023

Ausgelaufenes Öl aus Bohrplattformen und Tankern verursachen Umweltverschmutzungen und einen Biodiversitätsverlust. Forschende u.a. der Uni Stuttgart versetzen der Bewältigung von Ölkatastrophen durch Biotenside nun einen Schub nach vorn: Mit ihrer neusten Forschung konnten sie nachweisen, dass der mikrobiologische Abbau von Ölkomponenten im Meerwasser durch Biotenside gesteigert werden kann im Vergleich zu herkömmlichen Dispersionsmitteln.
-
Projekt AlbLavendel - 20.11.2023

Blau-violette, duftende Felder wie in der Provence sollen in Zukunft auch auf der Schwäbischen Alb ein normaler Anblick sein. Im Projekt AlbLavendel starteten die Universität Hohenheim, die Firma naturamus GmbH und die Deutschen Institute für Textil- und Faserforschung Denkendorf erste Untersuchungen zum regionalen Anbau von Lavendel sowie zur Produktion ätherischer Öle und Nutzung von Destillationsresten für die Herstellung von Textilfasern.
-
Klimaneutrale Kläranlagen durch patentierte Echtzeitanalytik - 08.11.2023

Die Abwasserindustrie ist für einen Ausstoß an Treibhausgas-Emissionen verantwortlich, der dem der globalen Luftfahrt entspricht. Das Start-up Variolytics ermöglicht mit einer weltweit ersten Echtzeitanalytik, Treibhausgase in Kläranlagen erheblich zu reduzieren. Mit patentierter Sensortechnik und KI-gestützter Prozessoptimierung erhalten Kommunen und Industrie einen Mehrfachnutzen: Neben Lachgas werden Energiekosten und Ressourcen eingespart.
-
Wasser 3.0: #detect|remove|reuse - 31.10.2023

Wir alle verschmutzen unser Wasser mit den Dingen unseres Alltags. Dabei sammeln sich Mikroplastik und Mikroschadstoffe in teils erheblichen Ausmaßen an und sind nur schwer wieder zu entfernen. Mit zunehmend verheerenden Folgen für Umwelt und Gesundheit. Das gemeinnützige Start-up Wasser 3.0 aus Karlsruhe hat ein Verfahren entwickelt, um diese Verschmutzungen maßgeschneidert aufzuspüren, zu entfernen und anschließend wiederverwerten zu können.










